程序1求解,目录和文件内容已展示
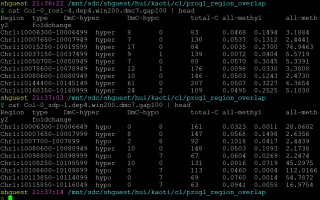
程序1: 两个文件区域的交集
/mnt/sdc/shguest/hui/kaoti/c1/prog1_region_overlap
计算两个*gap100 文件内第一列染色体和染色体起始位置-终止位置的overlap区域
区分第二列 hyper/hypo 计算两个*gap100 文件内第一列染色体和染色体起始位置-终止位置的overlap区域
【以下回答由 GPT 生成】
解决方案
对于给定的问题,我们可以使用Linux操作系统的一些工具和命令来解决。
首先,我们需要确保系统上安装了以下工具:
grep: 用于在文件中搜索指定的模式。awk: 用于文本处理和格式化。sort: 用于排序文件内容。uniq: 用于去除重复的行。
如果系统上没有安装这些工具,请根据您的Linux发行版,使用相应的包管理器进行安装(例如apt、yum等)。
接下来,我们将分步骤解决问题。
第一步: 计算两个gap100文件内第一列染色体和染色体起始位置-终止位置的overlap区域。
首先,我们可以使用grep命令从文件中提取第一列染色体和染色体起始位置-终止位置的内容。假设文件1是file1.txt,文件2是file2.txt,我们可以使用以下命令提取内容:
grep -o -P '^[^\t]+' file1.txt > file1_tmp.txt
grep -o -P '^[^\t]+' file2.txt > file2_tmp.txt
接下来,我们使用sort和uniq命令对提取的内容进行排序和去重,以获得两个文件之间的交集。我们将结果保存在overlap.txt中。
sort file1_tmp.txt file2_tmp.txt | uniq -d > overlap.txt
现在,overlap.txt文件中包含了两个文件之间的交集。
第二步: 区分第二列hyper/hypo,计算两个gap100文件内第一列染色体和染色体起始位置-终止位置的overlap区域。
为了区分第二列中的内容,我们可以使用awk命令来处理文件。
假设文件1是file1.txt,文件2是file2.txt,我们可以使用以下awk命令:
awk -F'\t' '{ print $1"\t"$2 }' file1.txt > file1_tmp.txt
awk -F'\t' '{ print $1"\t"$2 }' file2.txt > file2_tmp.txt
awk 'NR==FNR{a[$1,$2];next} ($1,$2) in a' file1_tmp.txt file2_tmp.txt > overlap.txt
以上命令将从文件中提取第一列和第二列的内容,并将结果保存在临时文件中。然后,使用awk命令比较两个文件中相同的记录,并将结果保存在overlap.txt中。
现在,overlap.txt文件中包含了两个文件之间第一列和第二列同时重复的记录。
以上就是解决问题的具体步骤和指导,希望能对您有所帮助。如果您有任何其他问题,请随时提问。
如果你已经解决了该问题, 非常希望你能够分享一下解决方案, 写成博客, 将相关链接放在评论区, 以帮助更多的人 ^-^