程序2求解,*.txt类型一样,要和结果gene.cov.xls一样
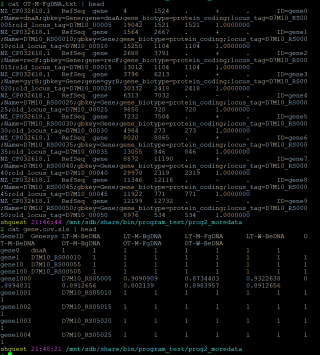
程序2:多样品覆盖度合并
/mnt/sdc/shguest/hui/kaoti/c2/prog2_moredata
合并*txt文件最后一列,并生成基因和样品的覆盖度矩阵,结果参考: gene.cov.xls
【以下回答由 GPT 生成】
我可以为您提供一个解决方案。以下是将多个.txt文件合并并生成与gene.cov.xls相同格式的覆盖度矩阵的步骤。
- 首先,您需要收集所有需要合并的.txt文件的路径。您可以使用Python的os模块来列出指定文件夹中的所有.txt文件路径。以下代码可实现该功能:
import os
folder_path = "<文件夹路径>"
file_paths = []
for file_name in os.listdir(folder_path):
if file_name.endswith(".txt"):
file_path = os.path.join(folder_path, file_name)
file_paths.append(file_path)
请将<文件夹路径>替换为您存储.txt文件的文件夹的路径。
- 然后,您需要将每个文件的最后一列提取出来,并将它们合并为覆盖度矩阵。您可以使用Python的pandas库来处理和合并数据。以下是实现该功能的代码:
import pandas as pd
# 创建一个空的DataFrame作为合并结果
merged_df = pd.DataFrame()
# 遍历每个文件路径进行处理和合并
for file_path in file_paths:
# 读取当前文件的数据
df = pd.read_csv(file_path, delimiter="\t")
# 提取最后一列数据
last_column = df.iloc[:, -1]
# 将数据添加到合并结果DataFrame的一个新列中
merged_df[file_path] = last_column
# 将合并结果DataFrame保存为gene.cov.xls文件
output_file_path = "<输出文件路径>"
merged_df.to_excel(output_file_path, index=False)
请将<输出文件路径>替换为您希望保存合并结果的文件路径。
- 最后,您可以使用终端或命令行运行程序2来生成覆盖度矩阵。使用以下命令行命令运行程序2:
/mnt/sdc/shguest/hui/kaoti/c2/prog2_moredata <输出文件路径>
请将<输出文件路径>替换为您在上一步中保存合并结果的文件路径。
这就是解决该问题的步骤。希望对您有所帮助。如果您有任何问题,请随时提问。
如果你已经解决了该问题, 非常希望你能够分享一下解决方案, 写成博客, 将相关链接放在评论区, 以帮助更多的人 ^-^