使用python将分子模拟输出.gro文件
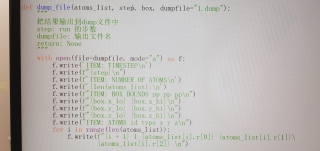
将这部分输出.dump代码改成输出.gro代码,测试可以用vmd导入
望采纳
首先,需要按照GRO文件的格式更改输出的内容。下面是一个示例GRO文件的格式:
System title
3 # 总原子数
1SOL OW1 1 1.00000 1.00000 1.00000
1SOL HW2 2 1.00000 1.00000 1.00000
1SOL HW3 3 1.00000 1.00000 1.00000
2.828427 1.414213 1.414213 # x, y, z方向的晶格长度(可选)
根据这个格式,可以更新代码如下:
def dump_file(atoms_list, step, box, dumpfile="1.gro"):
with open(file=dumpfile, mode="a") as f:
f.write("System title\n") # 标题
f.write(f"{len(atoms_list)} # 总原子数\n")
for i in range(len(atoms_list)):
# 每个原子的信息,包括编号、类型、坐标
f.write(f"1SOL OW{i + 1} {i + 1} {atoms_list[i].r[0]} {atoms_list[i].r[1]} {atoms_list[i].r[2]}\n")
f.write(f"{box.x_lo} {box.x_hi} {box.u_lo} {box.y_hi} {box.z_lo} {box.z_hi}\n") # 晶格长度
分子动力学模拟gro格式转换为 car
https://blog.csdn.net/weixin_50566616/article/details/119475100