生物python文本读写多条链的反向互补序列

在不使用re等函数的前提下完成,求大佬帮忙写一下,文本文件内容如下
https://www.cnblogs.com/yahengwang/p/9332561.html
# -*- coding: UTF-8 -*-
def Treated(Inputfile):
Transf={'A':'T',
'T':'A',
'G':'C',
'C':'G',
'a':'t',
't':'a',
'c':'g',
'g':'c'}
IN=open(Inputfile)
OUT=open("result.fas","w+")
for line in IN:
line=line.strip("\n")
if line.startswith('>'):
Writeline=line+"_rev\n"
OUT.write(Writeline)
else:
Bases=list(line)
Bases=[Transf[base] for base in Bases]
Writeline=''.join(Bases)
OUT.write(Writeline+"\n")
IN.close()
OUT.close()
if __name__ == "__main__":
Treated("GP.fas")
###################################################################################
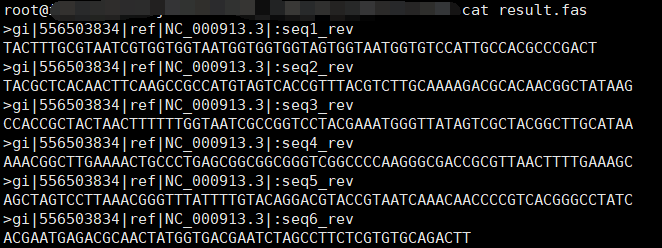
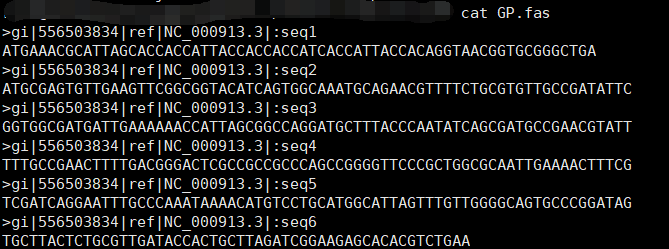
以下为文件示例:
输入文件示例:(GP.fas)
输出结果文件示例:(result.fas)