这样的图怎么做NMDS分析

有没有人会用R做NMDS分析,画这样的图啊。
找了代码:
#!/usr/bin/env Rscript
#install.packages("vegan") #安装vegan包
library(vegan)
sample <- read.table(file.choose(),sep = "\t",row.names = 1)#第一行为列名
group <- read.table(file.choose(),sep = "\t",header = T,row.names = 1)#其中header=T表示将文件中第一行设为列名字。row.names= 1表示第一列设为行名
group1 <- group[match(rownames(sample),rownames(group)),] #匹配行名,很重要group = group[rownames(sample),]
#nmds分析
nmds1 <- metaMDS(sample, distance = 'bray', k = 3)
summary(nmds1)
#提取数据
nmds1.stress <- nmds1$stress
nmds1.point <- data.frame(nmds1$point)
nmds1.species <- data.frame(nmds1$species)
sample_site <- nmds1.point[1:2]
sample_site$names <- rownames(sample_site)
colnames(sample_site)[1:2] <- c('NMDS1', 'NMDS2')
#合并分组数据
sample_site <- cbind(sample_site,group1)
#给分组排序
sample_site$Depth <- factor(sample_site$Depth,levels =c('0m','25m','50m','117m','150m', '200m',
ordered = FALSE ))
sample_site$Particlesize <- factor(sample_site$Particlesize, levels = c("0.2-0.6μm","0.6-1.2μm","1.2-2μm","2-20μm",">20μm",
ordered = FALSE))
View(sample_site)
#NMDS图绘制
windowsFonts(TNM = windowsFont("Times New Roman"))#设置字体
nmds_plot <- ggplot() +
geom_point(data = sample_site, aes(NMDS1, NMDS2,color = Particlesize, shape = Depth), size = 5, alpha = 0.8) + #可在这里修改点的透明度、大小
scale_shape_manual(values = c(15,17,18,19,20,79)) + #可在这里修改点的形状
scale_color_manual(values = c("#99CCFF","#FF9999","#FFCC33","#99CC99",
"#996699" )) + #可在这里修改点的颜色
scale_fill_manual(values = c("black" ),aesthetics = c("fill")) +#填充颜色
#geom_ord_ellipse(aes(sample_site$NMDS1,sample_site$NMDS2,group= sample_site$Particlesize),ellipse_pro = 0.8,linetype=2,size=0.7,color='firebrick')+##添加0.8置信椭圆
#geom_ord_ellipse(aes(sample_site$NMDS1,sample_site$NMDS2,group= sample_site$Particlesize,color= sample_site$Particlesize),ellipse_pro = 0.9,linetype=3,size=1)+ ##添加0.9置信椭圆
theme(panel.grid = element_blank(), panel.background = element_rect(color = 'black', fill = 'transparent')) + #去掉背景
theme(legend.key = element_rect(fill = 'transparent'), legend.text =element_text(family="TNM"), legend.title = element_blank()) + #去掉图例标题及标签背景
labs(x = 'Dimension1', y = 'Dimension2', title = paste('Stress =', round(nmds1$stress, 2))) +
theme(plot.title = element_text(hjust = 0.8,family="TNM",face = "bold"))+ #标题
theme(axis.text.x =element_text(family="TNM",face = "bold"),axis.text.y =element_text(family="TNM",face = "bold"),
legend.text = element_text(family="TNM"),panel.grid.minor = element_blank())+
theme(axis.title.y = element_text(family="TNM",face = "bold"),axis.title.x = element_text(family="TNM",face = "bold"))
#theme(panel.background = element_blank(),axis.line = element_line(color = "black"))#去上右边框
nmds_plot
总在分组排序的时候出现错误:
```r
Error in `$<-.data.frame`(`*tmp*`, Depth, value = integer(0)) :
replacement has 0 rows, data has 30
```
不知道你这个问题是否已经解决, 如果还没有解决的话:- 建议你看下这篇博客👉 :笔记—R语言NMDS
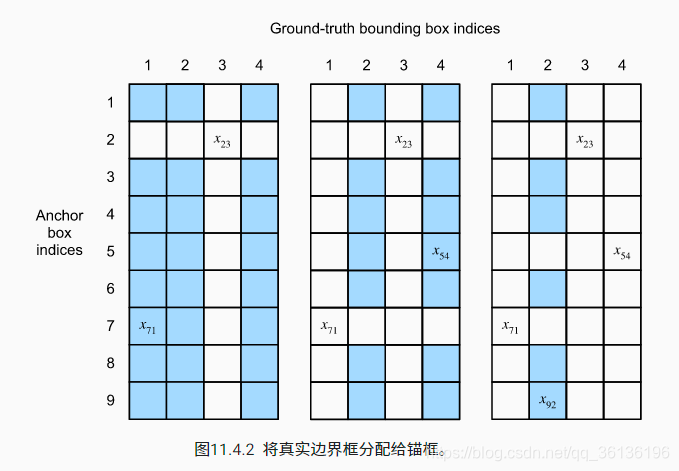
- 除此之外, 这篇博客: 锚框分配算法详解,偏移量代码详解,nms详解中的 锚框分配 部分也许能够解决你的问题, 你可以仔细阅读以下内容或者直接跳转源博客中阅读:
对于生成的锚框,计算得到任意一个锚框与所有的真实边界框的iou后,如何将真实边界框分配给锚框呢(也就是每一个锚框对应哪一个真实的边界框呢,或者对应背景呢),以下来自《动手学》

- 以下回答来自chatgpt:
回答:
可以在R中使用NMDS(Nonmetric Multidimensional Scaling)分析制作该类图像。以下是相关的R代码和指导:
- 首先,安装并加载相应的包:
install.packages("vegan") # 安装 vegan 包 library(vegan) # 加载 vegan 包- 接着,导入数据并进行排序:
# 假设数据文件名为 mydata.csv,第1列为物种名称(Species),第2-5列为物种的数量 mydata <- read.csv("mydata.csv", header = TRUE, row.names = 1) # 导入数据 mydata # 查看数据 # 对数据进行排序,例如按照物种数量(Freq)从高到低排序 mydata <- mydata[order(-mydata$Freq), ] # 降序排列 mydata # 查看排序后的数据- 进行NMDS分析并生成图像:
# 对数据进行NMDS分析 nmds <- metaMDS(mydata) # 绘制NMDS图像 plot(nmds, display = "sites", type = "n") # 先绘制空白图像 text(nmds, display = "sites", cex=0.7) # 标记物种名称 # 添加坐标轴标签 xlabel <- "NMDS 1" ylabel <- "NMDS 2" mtext(side=1, xlabel) mtext(side=2, ylabel) # 添加图例 legend("topright", legend = "Group", col = c("red","blue","green"),pch=19)以上是制作NMDS图像的基本步骤。在实际操作中,需要根据数据特点进行参数调整、美化图像等。
至于问题中提到的“遇到了一些错误”,由于未提供具体错误信息和代码,无法给出详细解决方法。建议在出现错误时先认真查看错误信息,结合代码进行排查,或在相关R社区或论坛寻求帮助。
如果你已经解决了该问题, 非常希望你能够分享一下解决方案, 写成博客, 将相关链接放在评论区, 以帮助更多的人 ^-^