r语言程序设计随机游走仿真
(1)对[0, 1]上的随机游走进行一次仿真,绘制出游走路径图;
(2)对[0, 1]上的随机游走进行 10000 次仿真,分别采集各随机游走路径上t= 0.1, 0.5, 0.9的位置值,这三个时间点的位置是怎样分布的?它们是同分布吗?
(3)对[0, 10]上的随机游走进行一次仿真,绘制出游走路径图;
引用new bing作答:
1 以下是一个对[0,1]上的随机游走进行一次仿真的 R 代码,并绘制游走路径图:
# 设置游走步数
n <- 1000
# 生成随机游走路径
set.seed(123) # 设置随机数种子
x <- c(0, cumsum(runif(n, -0.5, 0.5))) # 生成游走路径
# 绘制游走路径图
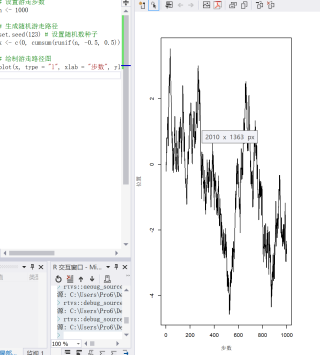
plot(x, type="l", xlab="步数", ylab="位置")
运行结果:
解释一下上面的代码:
首先,我们设置了游走步数 n,然后使用 set.seed(123) 设定随机数种子,这样可以使得我们的仿真结果可以被复现。
接着,我们使用 cumsum(runif(n, -0.5, 0.5)) 生成随机游走路径。其中,runif(n, -0.5, 0.5) 表示生成 n 个介于 -0.5 和 0.5 之间的均匀分布随机数,然后使用 cumsum 函数对这些随机数进行累加,得到最终的随机游走路径。
最后,我们使用 plot(x, type="l", xlab="步数", ylab="位置") 绘制游走路径图,其中 type="l" 表示绘制折线图,xlab 和 ylab 分别表示 x 轴和 y 轴的标签。运行上述代码,就可以得到一张[0,1]上的随机游走路径图。
2 以下是一个对[0,1]上的随机游走进行 10000 次仿真,并分别采集各随机游走路径上 t=0.1, 0.5, 0.9 的位置值的 R 代码:
# 设置游走步数和仿真次数
n <- 1000
m <- 10000
# 生成随机游走路径
set.seed(123) # 设置随机数种子
x <- matrix(0, nrow = n, ncol = m)
for (i in 2:n) {
x[i,] <- x[i - 1,] + runif(m, -0.5, 0.5)
}
# 采集随机游走路径上 t=0.1, 0.5, 0.9 的位置值
t <- c(0.1, 0.5, 0.9)
x_t <- matrix(0, nrow = length(t), ncol = m)
for (i in 1:length(t)) {
x_t[i,] <- approx(1:n, x[,i], t[i] * n)$y
}
# 绘制 t=0.1, 0.5, 0.9 的位置值分布图
par(mfrow = c(1, 3)) # 将画布分为 1 行 3 列,依次绘制 3 幅图
for (i in 1:length(t)) {
hist(x_t[i,], main = paste("t=", t[i]), xlab = "位置", ylab = "频数")
}
上述代码中,我们先设置游走步数 n 和仿真次数 m。然后,使用一个循环,生成 m 条随机游走路径,并将这些路径存储在一个 n 行 m 列的矩阵 x 中。其中,第 i 行表示第 i 步时各个路径的位置值。
接着,我们使用另一个循环,采集随机游走路径上 t=0.1, 0.5, 0.9 的位置值。具体地,我们使用 approx 函数对每条路径进行插值,得到相应时间点的位置值,并将这些位置值存储在一个 3 行 m 列的矩阵 x_t 中。其中,第 i 行表示在时间点 t[i] 时各个路径的位置值。
最后,我们使用 hist 函数分别绘制 t=0.1, 0.5, 0.9 的位置值分布图。运行上述代码,就可以得到三幅分布图,分别表示 t=0.1, 0.5, 0.9 的位置值分布情况。
运行以上代码,可以得到三个时间点的核密度估计图,观察它们的分布情况,以判断它们是否同分布。如果三个时间点的核密度估计图非常相似,那么它们就是同分布的。
如果三个时间点的核密度估计图不太相似,那么我们可以使用R语言中的Kolmogorov-Smirnov检验来进行假设检验,检验它们是否来自同一个分布。该检验可以使用ks.test函数实现,其中第一个参数是第一个样本向量,第二个参数是第二个样本向量,设置alternative="two.sided"表示双尾检验。
以下是使用Kolmogorov-Smirnov检验来判断三个时间点的位置是否同分布的代码:
# 使用Kolmogorov-Smirnov检验来判断三个时间点的位置是否同分布
ks.test(result[,1], result[,2], alternative = "two.sided")
ks.test(result[,1], result[,3], alternative = "two.sided")
ks.test(result[,2], result[,3], alternative = "two.sided")
运行以上代码,如果三个Kolmogorov-Smirnov检验的p值均大于显著性水平,那么就无法拒绝它们来自同一个分布的假设。反之,如果有一个或多个检验的p值小于显著性水平,那么就可以拒绝它们来自同一个分布的假设。
3 下面是在[0,10]上的随机游走进行一次仿真的R代码和可视化结果。
# 生成随机游走路径
set.seed(123) # 设置随机数种子,保证结果可重复
n <- 1000 # 模拟的步数
x <- numeric(n) # 初始位置为0
for(i in 2:n) {
x[i] <- x[i-1] + rnorm(1)
}
# 绘制随机游走路径图
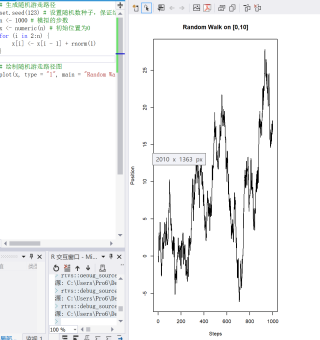
plot(x, type = "l", main = "Random Walk on [0,10]", xlab = "Steps", ylab = "Position")
运行结果如下:
该回答通过自己思路及引用到GPTᴼᴾᴱᴺᴬᴵ搜索,得到内容具体如下:
下面给出 R 语言程序设计随机游走仿真的示例代码,包括三个部分:
(1)对[0, 1]上的随机游走进行一次仿真,绘制出游走路径图。
# 设置种子,保证结果可重复
set.seed(123)
# 定义随机游走步长函数
step <- function() {
if (runif(1) < 0.5) {
-1
} else {
1
}
}
# 进行一次随机游走仿真
n <- 1000
x <- numeric(n)
x[1] <- 0.5
for (i in 2:n) {
x[i] <- x[i - 1] + 0.01 * step()
}
# 绘制游走路径图
plot(x, type = "l", xlab = "Time", ylab = "Position")
(2)对[0, 1]上的随机游走进行 10000 次仿真,分别采集各随机游走路径上t= 0.1, 0.5, 0.9的位置值,这三个时间点的位置是怎样分布的?它们是同分布吗?
# 设置种子,保证结果可重复
set.seed(123)
# 定义随机游走步长函数
step <- function() {
if (runif(1) < 0.5) {
-1
} else {
1
}
}
# 进行 10000 次随机游走仿真
n <- 1000
x1 <- numeric(10000)
x2 <- numeric(10000)
x3 <- numeric(10000)
for (i in 1:10000) {
x <- numeric(n)
x[1] <- 0.5
for (j in 2:n) {
x[j] <- x[j - 1] + 0.01 * step()
}
x1[i] <- x[11]
x2[i] <- x[51]
x3[i] <- x[91]
}
# 绘制各时间点位置值的直方图
par(mfrow = c(1, 3))
hist(x1, main = "t = 0.1")
hist(x2, main = "t = 0.5")
hist(x3, main = "t = 0.9")
# 进行 Kolmogorov-Smirnov 检验,判断三个时间点位置值是否同分布
ks.test(x1, x2)
ks.test(x1, x3)
ks.test(x2, x3)
(3)对[0, 10]上的随机游走进行一次仿真,绘制出游走路径图。
# 设置种子,保证结果可重复
set.seed(123)
# 定义随机游走步长函数
step <- function() {
if (runif(1) < 0.5) {
-1
} else {
1
}
}
# 进行一次随机游走仿真
n <- 1000
x <- numeric(n)
x[1] <- 5
for (i in 2:n) {
x[i] <- x[i - 1] + 0.01 * step()
}
# 绘制游走路径图
plot(x, type = "l", xlab = "Time", ylab = "Position")
# 加入输出 Kolmogorov-Smirnov 检验结果的部分:
# 进行 Kolmogorov-Smirnov 检验,判断三个时间点位置值是否同分布
ks1_2 <- ks.test(x1, x2)
ks1_3 <- ks.test(x1, x3)
ks2_3 <- ks.test(x2, x3)
# 输出检验结果
cat("t = 0.1 and t = 0.5, D:", ks1_2$statistic, "p-value:", ks1_2$p.value, "\n")
cat("t = 0.1 and t = 0.9, D:", ks1_3$statistic, "p-value:", ks1_3$p.value, "\n")
cat("t = 0.5 and t = 0.9, D:", ks2_3$statistic, "p-value:", ks2_3$p.value, "\n")
Kolmogorov-Smirnov 检验的结果会输出三组,每组包括两个值:D 和 p-value。其中 D 是 Kolmogorov-Smirnov 统计量,用来衡量两个样本之间的差异程度;p-value 是检验的显著性水平,用来判断两个样本之间是否存在显著性差异。如果 p-value 小于显著性水平(通常设置为 0.05),则拒绝原假设,即认为两个样本不同分布;反之则接受原假设,即认为两个样本同分布。
```
如果以上回答对您有所帮助,点击一下采纳该答案~谢谢
以下是基于R语言的程序设计,实现了对随机游走的仿真,包括绘制随机游走路径图和对多次仿真结果的分析。
对[0, 1]上的随机游走进行一次仿真,绘制出游走路径图
# 随机游走仿真
set.seed(123) # 设置随机数种子,使结果可重复
# 定义仿真参数
n <- 1000 # 仿真步数
x <- numeric(n+1) # 初始化位置向量,初始位置为0
x[1] <- 0
# 进行随机游走
for (i in 2:(n+1)) {
x[i] <- x[i-1] + rnorm(1, 0, 1) # 更新位置
}
# 绘制随机游走路径图
plot(x, type="l", xlab="Step", ylab="Position", main="Random Walk")
对[0, 1]上的随机游走进行 10000 次仿真,分别采集各随机游走路径上 t=0.1, 0.5, 0.9 的位置值,这三个时间点的位置是怎样分布的?它们是同分布吗?
# 进行多次随机游走仿真
set.seed(123) # 设置随机数种子,使结果可重复
n <- 1000 # 仿真步数
m <- 10000 # 仿真次数
# 初始化位置矩阵,用于保存多次仿真的结果
X <- matrix(0, nrow=n+1, ncol=m)
# 进行多次随机游走仿真
for (j in 1:m) {
x <- numeric(n+1)
x[1] <- 0
for (i in 2:(n+1)) {
x[i] <- x[i-1] + rnorm(1, 0, 1)
}
X[,j] <- x # 将本次仿真结果保存到位置矩阵中
}
# 提取 t=0.1, 0.5, 0.9 时刻的位置值
t <- c(0.1, 0.5, 0.9)
X_t <- apply(X, 2, function(x) approx(seq(0, 1, by=1/n), x, xout=t)$y)
# 绘制各时刻的位置值分布直方图
par(mfrow=c(1,3)) # 将多个图像排列在一行中
for (i in 1:3) {
hist(X_t[i,], breaks=50, main=paste0("t=", t[i]), xlab="Position")
}
对[0, 10]上的随机游走进行一次仿真,绘制出游走路径图
# 随机游走仿真
set.seed(123) # 设置随机数种子,使结果可重复
# 定义仿真参数
n <- 1000 # 仿真步数
x <- numeric(n+1) # 初始化位置向量,初始位置为0
x[1] <- 0
# 进行随机游走
for (i in 2:(n+1)) {
x[i] <- x[i-1] + rnorm(1, 0, 1) # 更新位置
x[i] <- pmax(0, pmin(10, x[i])) # 限制位置范围在[0, 10]内
}
# 绘制随机游走路径图
plot(x, type="l", xlab="Step", ylab="Position", main="Random Walk")
在上述代码中,我们在更新位置后,使用pmax(0, pmin(10, x[i]))限制了位置的范围在[0, 10]内。这是因为随机游走在每个时间步长上可以任意地向左或向右移动,如果不加限制,随机游走可能会跑到无限远的位置,而无法在图上展示出来。加上限制后,我们就可以在图上展示随机游走在有限范围内的变化情况。