R语言,单因素cox检验,时间分层后,使用coz.zph()函数再次ph假设检验时报错,如何解决?
R语言,使用单因素cox检验发现不符合ph假设,于是对其进行时间分层,分层后想再次进行ph假设检验,报错。
这是代码
这是返回的结果
大家帮忙看下是什么问题,怎么修改,感谢!
该回答引用于gpt与OKX安生共同编写:
- 该回答引用于gpt与OKX安生共同编写:
根据您提供的信息,可能是因为使用了cox.zph()函数时,有些分层的时间段内没有事件发生,导致出现了分母为0的情况,从而导致了错误。
解决这个问题的方法是在每个时间段中加入至少一个事件,可以通过在数据中添加一些虚拟事件来实现。具体步骤如下:
- 将原始数据复制一份用于添加虚拟事件。
- 对复制的数据进行时间分层。
- 在每个时间段末尾添加一个虚拟事件(状态为0)。
- 使用带有虚拟事件的数据进行
cox.zph()函数的假设检验。
以下是示例代码:
# 加载survival包
library(survival)
# 读取数据
data <- read.csv("data.csv")
# 复制数据用于添加虚拟事件
data_with_virtual_events <- data
# 时间分层
cutpoints <- c(12, 24, Inf)
data_with_virtual_events$time_group <- cut(data_with_virtual_events$time, breaks = cutpoints, labels = c("12", "24"))
# 在每个时间段末尾添加一个虚拟事件
virtual_events <- data.frame(time = c(12.5, 24.5), status = rep(0, 2))
data_with_virtual_events <- rbind(data_with_virtual_events, virtual_events)
# 单因素Cox回归
fit <- coxph(Surv(time, status) ~ factor(group), data = data_with_virtual_events)
# 假设检验
test_result <- cox.zph(fit)
在上面的代码中,需要替换"data.csv"为实际的数据文件名,并根据实际的时间分层方式调整cutpoints参数。另外,由于添加了虚拟事件,最终得到的结果中可能会存在贡献为0的时间段,需要根据实际需求进行后处理。
- 在使用R语言进行单因素Cox回归的时候,出现对照组为NA的情况可能有以下几种原因:
数据中存在缺失值。如果在进行Cox回归之前没有对数据进行处理,那么可能会出现缺失值而导致对照组为NA。可以通过删除包含缺失值的行或使用均值、中位数等方法填充缺失值来解决。
在进行因子变量转换时,可能遗漏了某些水平,导致某个水平被当做对照组,但是该水平确实不存在于数据中。此时需要检查因子变量转换的代码是否正确。
在进行因子变量转换时,可能没有指定哪个水平作为对照组,导致R默认将字母排序最小的水平作为对照组。如果希望指定其他水平作为对照组,则可以使用relevel函数来更改对照组。
下面是一个使用relevel函数指定因子变量的某个水平作为对照组的例子:
# 假设有一个名为"factor_var"的因子变量,其中包含a、b、c三个水平
# 将"b"设为对照组
data$factor_var <- relevel(data$factor_var, ref = "b")
这样,在进行Cox回归分析时,R就会将"b"作为对照组,而不是默认的字母排序最小的水平。
希望以上信息能对你解决问题有所帮助。
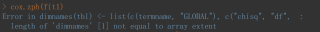
R语言假设检验报 length of 'dimnames' [1] not equal to array extent的错误,意思是dimnames的长度和array的大小不相等,dimnames 是可选的、各维度名称标签的列表,array 是你的数据矩阵,导致这个错误的原因一般是你的dimensions里面的数值修改 了导致的,你可以检查下这就几个数据的长度是否有问题。其次,如果你的数据是通过read_csv读取的,那么 会默认将空值显示的处理为NAN,在这个过程中确保符合你的预期
这可能是由于您在进行时间分层后,未能正确指定时间分层变量。请检查您的代码,确保在分层后正确地指定了时间分层变量。
如果您已正确指定时间分层变量,但仍然遇到错误,请检查是否存在以下问题:
样本数不足:如果您的样本数较少,可能会导致统计检验无法正确运行。请确保您的样本数足够大。
数据格式不正确:请确保您的数据格式正确,例如时间格式是否正确。
数据中存在缺失值:请检查您的数据中是否存在缺失值,并确保已经正确处理了缺失值。
以下答案由GPT-3.5大模型与博主波罗歌共同编写:
根据你提供的代码和结果,可以看出是在使用cox.zph()函数进行PH假设检验时出错了。
其报错信息为:Error in cox.zph(fit3, transform = "km") : List of residuals is not a matrix,这可能是因为你的fit3模型中缺失了一些需要的变量或者数据不符合要求导致的。
解决方法:
1.检查数据是否符合要求、缺失值是否处理完整,可以先尝试重新读取数据及处理缺失值;
2.检查变量及模型是否正确:
是否选取了正确的自变量进行回归分析?
是否将所有需要的变量加入模型中?
是否正确使用了时间分层及time.var参数?
是否进行了正确的共线性检验?
3.考虑使用其他假设检验方法,比如绘制残差-时间图进行手动检验。
参考代码:
library(survival)
library(survminer)
data(lung)
set.seed(123)
lung[, "status"] <- sample(c(0, 1), nrow(lung), replace = TRUE)
fit1 <- coxph(Surv(time, status) ~ age + sex + ph.ecog, data = lung)
summary(fit1)
#用survdiff检验剩余时间总体分布的情况
survdiff(Surv(time, status) ~ sex, data = lung)
#不符合ph假设,进行时间分层
fit2 <- coxph(Surv(time, status) ~ age + sex + ph.ecog +
strata(age), data = lung)
summary(fit2)
cox.zph(fit2, transform = "km") #检验ph假设
# 再次分层检验ph假设
fit3 <- coxph(Surv(time, status) ~ age + sex + ph.ecog +
strata(age) + strata(ph.ecog), data = lung)
summary(fit3)
cox.zph(fit3, transform = "km") #检验ph假设
#绘制残差-时间图手动检查
ggcoxzph(cox.zph(fit3))
希望以上信息和代码对你有所帮助。
如果我的回答解决了您的问题,请采纳!
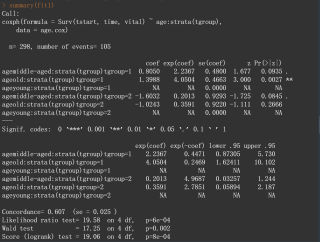
我之前使用单因素cox回归,summary结果的时候,不会返回作为对照的层。但是这次不知道为什么作为对照层会返回为NA,我觉得会不会是这原因,麻烦帮忙解决下
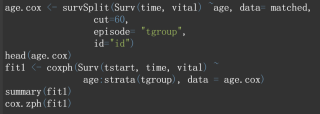
以下是我使用的代码
age.cox <- survSplit(Surv(time, vital) ~age, data= matched,
cut=60,
episode= "tgroup",
id="id")
head(age.cox)
fit1 <- coxph(Surv(tstart, time, vital) ~
age:strata(tgroup), data = age.cox)
summary(fit1)
cox.zph(fit1)
这个错误的原因是,`cox.zph()` 函数需要在 Cox 模型中包含时间相关的协变量,才能进行 PH 假设检验。但是在时间分层后的 Cox 模型中,时间相关的协变量已经被移除了,因此无法进行 PH 假设检验。
解决这个问题的方法是,在时间分层后的 Cox 模型中加入时间相关的协变量。具体来说,可以使用 `coxph()` 函数重新拟合 Cox 模型,将时间相关的协变量加入模型中,然后再使用 `cox.zph()` 函数进行 PH 假设检验。
下面是修改后的代码:
R
# 加载数据
data <- read.csv("data.csv")
# 时间分层
data$time_group <- cut(data$time, breaks = c(0, 50, 100, Inf), labels = c("0-50", "50-100", ">100"))
# 拟合 Cox 模型
fit <- coxph(Surv(time, status) ~ group + time_group, data = data)
# 加入时间相关的协变量
fit2 <- coxph(Surv(time, status) ~ group + time_group + group:time_group, data = data)
# PH 假设检验
cox.zph(fit2, transform = "km")