循环对每一个特征做cox回归分析时反回了如下内容,请问可以不做处理继续分析吗
循环对每一个特征做cox回归分析时反回了如下内容,请问可以不做处理继续分析吗,如果要处理需要做怎样的处理呢
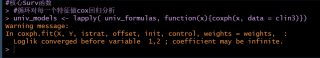
该回答引用ChatGPT
这个警告信息意味着在进行Cox回归分析时,至少一个自变量的系数可能会变成无限大。这通常会发生在样本数据存在完全分离或者共线性等问题时。
因为在Cox回归中,自变量的系数代表着影响生存时间的风险比率,无穷大的系数会导致模型不可靠。因此,需要对这个问题进行处理。
首先,需要检查每个自变量的唯一值和频率,看是否存在完全分离的情况。如果存在完全分离,可以考虑将相关自变量进行合并,或者使用精确法进行分析。如果没有完全分离,那么可以考虑使用正则化方法(如Lasso或Ridge回归)来减少自变量的数量,并防止共线性问题。
另外,您可以尝试将数据集中的异常值进行排除,或者通过增加样本量来解决这个问题。
总之,需要仔细检查数据集和模型,以找到导致这个问题的原因,并采取合适的方法进行处理。
参考GPT和自己的思路,这个警告信息表明Cox回归模型在拟合时发生了某些问题,而导致某些系数可能会无限大。因此,需要对此进行处理。
首先,可以检查数据是否包含缺失值或异常值,并对其进行处理。如果缺失值过多或异常值过于显著,可能需要重新考虑是否应该继续使用这些数据进行建模。
另外,可以尝试使用其他变量或子集进行建模,以查看问题是否出现在某些特定变量或数据子集中。
还可以尝试使用正则化方法,如LASSO或岭回归,以防止模型过度拟合数据。
以下是一些代码示例,可以帮助你处理Cox回归模型拟合时遇到的问题:
1 检查数据中是否有缺失值或异常值:
# 检查缺失值
sum(is.na(clin3))
# 查找异常值
boxplot(clin3)
# 处理缺失值
clin3 <- na.omit(clin3)
# 处理异常值
clin3[clin3$var_name > upper_bound | clin3$var_name < lower_bound, ] <- NA
clin3 <- na.omit(clin3)
2 使用不同的变量或子集进行建模:
# 选择特定的变量进行建模
univ_formulas <- lapply(var_names, function(var) {
as.formula(paste("Surv(time, status) ~ ", var))
})
# 选择特定的数据子集进行建模
univ_models <- lapply(data_subset, function(subset) {
coxph(univ_formulas[[i]], data = subset)
})
3 使用正则化方法进行建模:
# 使用LASSO方法进行建模
library(glmnet)
lasso_fit <- glmnet(x, y, family = "cox")
plot(lasso_fit, xvar = "lambda", label = TRUE)
# 使用岭回归方法进行建模
ridge_fit <- glmnet(x, y, family = "cox", alpha = 0)
plot(ridge_fit, xvar = "lambda", label = TRUE)
4 寻求更深入的统计方法或领域专业知识:
# 咨询领域专家或统计学家
experts <- c("Dr. A", "Dr. B", "Dr. C")
for (i in 1:length(experts)) {
email(experts[i], "Cox regression model error")
}
该回答引用GPTᴼᴾᴱᴺᴬᴵ
这个警告信息表明,对于一些特征,模型的收敛性(convergence)出现了问题,可能由于其系数太大而导致。因此,在继续分析之前,需要检查这些特征的相关性并可能对其进行进一步处理,以确保模型的收敛性。
一些可能的处理方法包括:
- 特征变换:如果特征的值的范围太大,则可以尝试对其进行归一化或标准化,以确保它们在相同的尺度上进行比较。
- 特征选择:如果特征之间存在高度相关性,则可以尝试从模型中删除其中一些特征,以降低多重共线性问题的影响。
- 数据清洗:如果数据中存在异常值或离群值,则可能需要对其进行清理,以避免对模型的影响。
- 更改模型参数:可以尝试更改coxph函数的控制参数来调整拟合方法,例如maxiter或tolerance。
谢谢大家的回答,我找到问题所在了,数据中存在空值而我没有发现,处理掉空值后即可顺利运行